検出の特異性
ほとんどの場合、プライマー設計の目的は、PCR の特異性を最大化することです。これは、多かれ少なかれ予測可能な多くの変数の影響によって決まります。重要な変数の 1 つは、プライマーの 3' 末端の配列です。
重要なのは、特異性を重視して設計された PCR アッセイは、非特異的な増幅産物を生成しないため、PCR 試薬と競合したり、主要な増幅反応を阻害したりすることがないため、広いダイナミック レンジにわたって高い効率を維持できる可能性が高くなります。
もちろん、場合によっては、特異性が最も重要ではない場合もあります。たとえば、密接に関連しているが異なる病原体を定量化することが目的の場合、特別な設計、最適化、および検証基準が必要になります。
融解曲線は、少なくとも単一の標的を増幅するかどうかの観点から、アンプリコンの特異性を評価するための標準的な方法です。ただし、融解曲線は、最適ではないプライマーと低いテンプレート濃度の複合効果によって影響を受ける可能性があるため、誤解を招く可能性があることを強調しなければなりません。
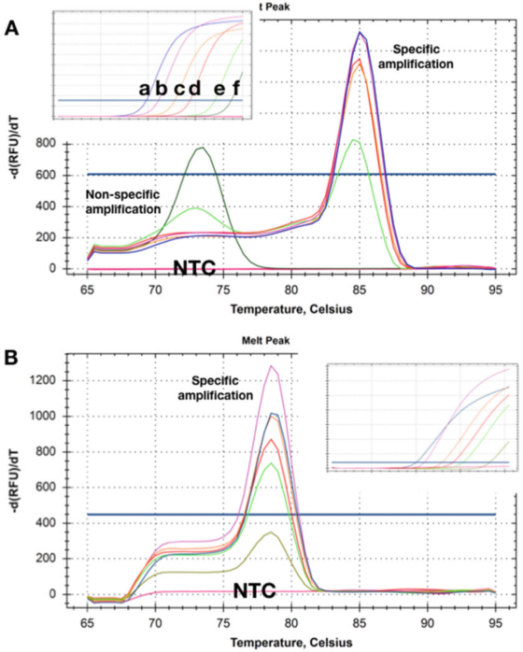
P5 |融解曲線は、異なる量の 2 つのターゲット DNA の 2 回の検出から得られた Tm シフトを示します。
A. より高い濃度 (ad)) では、qPCR 測定の完了後に明らかなプライマー二量体は存在しません。テンプレート濃度が 50 コピーに減少すると (e)、非特異的生成物が現れ始め、最低濃度では唯一の生成物になります (f)。
B. このテストでは、すべての標的濃度で同じ Tms が記録され、最低濃度 (5 コピー) でも明らかなプライマー二量体は存在しませんでした。これら 2 つの検出方法を使用した場合、NTC では増幅産物は検出されませんでした。
P5 は、テンプレートが異なる濃度で存在するサンプルで得られた溶解曲線を示しています。P5aは、2つの最も低い濃度において、生成された非特異的増幅産物のTmが特異的アンプリコンのTmよりも低いことを示している。
明らかに、この検出方法は、低濃度で存在するターゲットを検出するためには確実に使用できません。
興味深いことに、NTC、つまり DNA をまったく含まないサンプルは (非特異的) 増幅産物を記録せず、バックグラウンドのゲノム DNA が非特異的増幅/重合に関与する可能性があることを示しています。
場合によっては、このようなバックグラウンドプライマーや非特異的増幅を修正できない場合もありますが、多くの場合、どのようなテンプレート濃度でも NTC においても非特異的増幅が起こらない検出方法を設計することが可能です (P 5b)。
ここでは、Cq 35 でターゲット濃度の増幅を記録するだけでも、特異的な溶解曲線が生成されます。同様に、NTC は非特異的増幅の兆候を示さなかった。場合によっては、検出挙動が母液に依存し、特定の緩衝液組成では非特異的増幅のみが検出される場合があり、これは異なる Mg2+ 濃度に関連している可能性があります。
検出の安定性
Ta の最適化は、qPCR 検出の経験的検証および最適化プロセスにおける有用なステップです。NTC を増幅せずに最低の Cq を生成する温度 (または温度範囲) を示すことで、プライマー セットの堅牢性を直接示します。
感度の 2 ~ 4 倍の差は、mRNA 発現が高い人にとっては重要ではないかもしれませんが、診断検査では、陽性結果と偽陰性結果の差を意味する可能性があります。
qPCR プライマーの Ta 特性は大きく異なる場合があります。一部のテストはあまり堅牢ではなく、プライマーの最適な Ta 値の下で実行されないと、すぐに機能不全に陥ります。
このタイプの検出は現実世界では問題が発生することが多く、サンプルの純度、DNA の濃度、または他の DNA の存在が最適ではない可能性があるため、これは重要です。
さらに、標的コピー数は広範囲に変化する可能性があり、試薬、プラスチック製の器具、または機器は、テストの設定時に使用されるものと異なる場合があります。
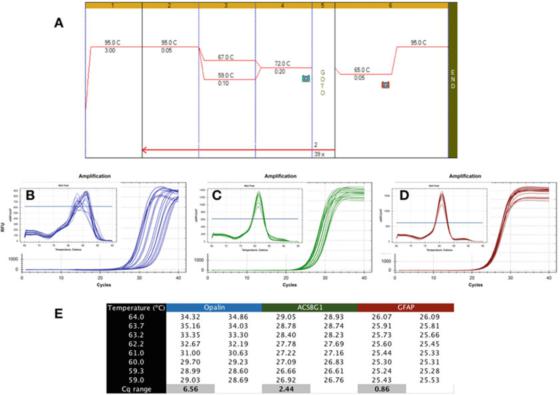
P6|温度勾配は、PCR 検出の堅牢性の違いを示します。
A. Bioline の Sensifast SYBR マスターミックス (カタログ番号 BIO-98050) を使用して、ヒト脳 RNA から調製した cDNA に対して PCR を実行します。
B. Bio-Rad の CFX qPCR 装置を使用して、アパレン (NM_033207、F: GCCATGGAGGAAAGTGACAGACC、R: CTCATGTGTGGGTGATCTCCTAGG) の増幅マップと溶解曲線を記録します。
C. ACSBG1 の増幅グラフと融解曲線 (NM_015162.4、F: CTACACTTCCGGCACCACTGG、R: GTCCACGTGATATTGTCTTGACTCAG)。
D. GFAP の増幅グラフと溶解曲線 (NM_002055.5、F: TGGAGAGGAAGATTGAGTCGCTGG、R: CGAACCTCCTCCTCGTGGATCTTC)。
E. 異なるアニーリング温度で記録された Cqs。7℃の温度勾配下で記録された Cq の違いを示します。
P6は、3つのヒト脳特異的遺伝子のプライマーを使用し、59℃と67℃の間の勾配Tas(P6a)を使用してqPCRが実行された、望ましくないテストの典型的な結果を示しています。
増幅グラフから、Opalin プライマーは最適な Ta 範囲が非常に狭いため (図 6b)、つまり Cq が広く分散しており、その結果、Cq が最適な Cqs Low と大幅に比較されるため、理想からはほど遠いことがわかります。
この検出方法は不安定であり、最適以下の増幅を引き起こす可能性があります。したがって、このプライマーのペアを再設計する必要があります。さらに、融解曲線分析 (挿入図) は、各 Ta の融解曲線が異なるため、この検出方法の特異性にも問題がある可能性があることを示しています。
P 6c に示されている ACSBG1 検出方法は、上記の Opalin 検出方法よりも堅牢ですが、依然として理想には程遠いため、改善できる可能性があります。
ただし、この検出方法によって生成された溶解曲線はすべての Ta (挿入図) で同じピーク値を示すため、堅牢性と特異性の間に必ずしも関連性がないことを強調します。
一方、ロバスト性テストははるかに耐性があり、P 6d に示されている GFAP テストのように、広範囲の Tas で同様の Cq が生成されます。
同じ摂氏 8 度の範囲で得られた Cqs の差は 1 未満であり、溶解曲線 (挿入図) はこの温度範囲での検出特性を確認しています。計算された Tas と実際の Ta 範囲は大きく異なる可能性があることに注意してください。
研究者が効率的なプライマーを設計するのに役立つように設計されたガイドラインは数多くありますが、そのほとんどは長年確立されてきたルールに基づいており、プライマーの 3' 末端に多くの注意が払われています。多くの場合、3' 末端に G または C を含め、2 つの G または C 塩基 (GC クランプ) を含めることが推奨されますが、最後の 5 塩基のうち 2 つを超えないようにします。
実際には、これらのルールは研究者を導くことができますが、すべての状況において必ずしも正しいとは限りません。
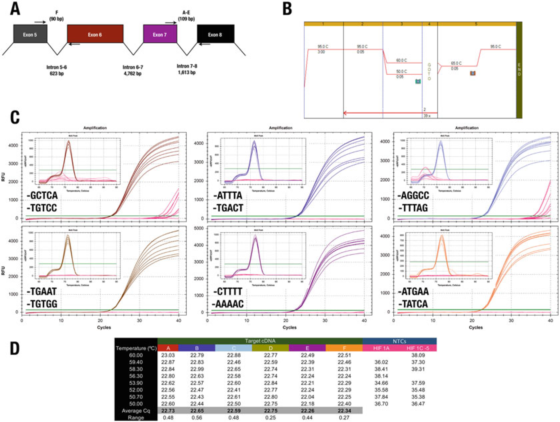
P7 |プライマーの 3' 末端は、特異性や効率にほとんど影響を与えません。
A. ヒト HIF-1α (NM_181054.2) 遺伝子のプライマーの位置。
B. Agilent Brilliant III SYBR Green 母液 (カタログ番号 600882) を使用して 6 つのテスト項目を増幅します。
C. Bio-Rad の CFX qPCR 機器および 3' 末端プライマーによって記録された増幅グラフおよび融解曲線。NTC は赤色で表示されます。
D. 各試験項目のCqs記録
たとえば、P 7 の結果は 3'end ルールに矛盾します。すべてのデザインは基本的に同じ結果をもたらしますが、NTC では 2 つのプライマーの組み合わせだけが非特異的増幅を引き起こします。
ただし、この場合、最大 30 塩基として A または T を使用しても特異性は低下しないため、GC クリップの効果はサポートできません。
F プライマーが GGCC で終わるテスト C では、NTC で Cq が記録されました。これは、30 末端でこれらの配列を回避する必要がある可能性があることを示しています。プライマーペアの最適な 3' 末端配列を決定する唯一の方法は、いくつかの候補プライマーを実験的に評価することであることを強調します。
増幅効率
重要なのは、非特異的 PCR 検出は決して特異的になることはありませんが、酵素、母液、添加剤、サイクリング条件を変更することで、さまざまな方法で増幅効率を調整し、最大化することができるということです。
PCR の検出効率を評価するには、標的核酸を 10 倍または 5 倍に段階希釈する「標準曲線法」を使用するのが最適です。
PCR アンプリコンまたは合成 DNA ターゲットを使用して標準曲線を作成する場合、これらのターゲットの段階希釈物を一定量のバックグラウンド DNA (ゲノム DNA など) と混合する必要があります。
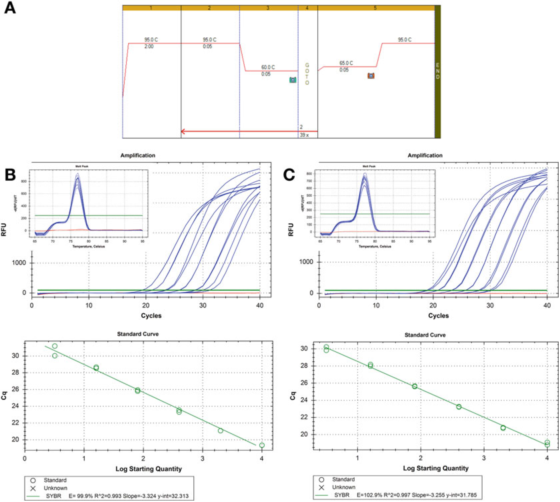
P8 |PCR の効率を評価するための希釈曲線。
A. HIF-1 のプライマー: F: AAGAACTTTTAGGCCGCTCA および R: TGTCCTGTGGTGACTTGTCC および Agilent の Brilliant III SYBR Green マスターミックス (カタログ番号 600882) を PCR および融解曲線条件に使用します。
B. 100 ng RNA を逆転写し、2 倍に希釈し、連続希釈した cDNA サンプルを 5 倍に希釈して 1 ng ヒトゲノム DNA にしました。融解曲線を挿入図に示します。
C. 2 番目の cDNA サンプルに対して RT 反応、希釈、段階希釈を繰り返しましたが、結果は同様でした。
P8は、2つの異なるcDNAサンプルに対して同じ検出方法を使用した2つの標準曲線を示しています。結果は同じ効率、約100%であり、R2値も類似しています。つまり、実験データと回帰直線またはデータの直線性の程度の間の適合度です。
2 つの標準曲線は比較可能ですが、まったく同じではありません。目的がターゲットを正確に定量化することである場合、不確実性を説明せずにコピー数の計算を行うことは受け入れられないことに注意する必要があります。
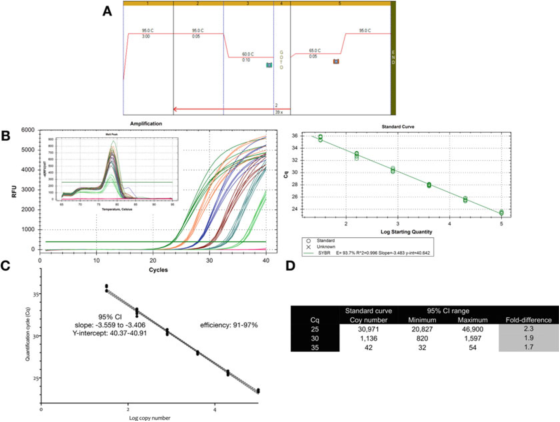
P9 |標準曲線を使用した定量化に伴う測定の不確かさ.
A. GAPDH (NM_002046) 用のプライマーを使用して、PCR および融解曲線条件を実行します。F: ACAGTTGCCATGTAGACC および R: TAACTGGTTGAGCACAGG および Bioline の Sensifast SYBR マスターミックス (カタログ番号 BIO-98050)。
B. Bio-Rad の CFX qPCR 装置で記録された増幅チャート、融解曲線、および標準曲線。
C. 標準曲線グラフと 95% 信頼区間 (CI)。
D. 希釈曲線から導出された 3 つの Cq 値のコピー数と 95% 信頼区間。
P9 は、最適化されたテストの場合、単一標準曲線の固有の変動が約 2 倍 (95% 信頼区間、最小値から最大値まで) であることを示しており、これは予想される最小の変動である可能性があります。
関連商品:
投稿時間: 2021 年 9 月 30 日