- PCR は、少量の DNA テンプレートから DNA を増幅するために使用される方法です。RT-PCR では、逆転写を使用して RNA ソースから DNA テンプレートを生成し、それを増幅します。
- PCR および RT-PCR は通常、エンドポイント反応ですが、qPCR および RT-qPCR は、PCR 反応中の生成物合成速度の速度論を使用して、存在するテンプレートの量を定量します。
- デジタル PCR などの新しい方法では、最初の DNA テンプレートの絶対定量が可能ですが、等温 PCR などの方法では、信頼性の高い結果を得るために高価な機器の必要性が軽減されます。
ポリメラーゼ連鎖反応 (PCR) は、DNA および RNA 配列を増幅および検出する比較的単純で広く使用されている分子生物学の手法です。多くの場合数日かかる従来の DNA クローニングと増幅の方法と比較して、PCR には数時間しかかかりません。PCR は感度が高く、特定の配列の検出と増幅に必要なテンプレートは最小限です。基本的な PCR 法は、単純な DNA および RNA の検出からさらに進歩しました。以下に、お客様の研究ニーズに合わせて Enzo Life Sciences で提供するさまざまな PCR 方法と試薬の概要を示します。私たちは、科学者が次の研究プロジェクトで使用する PCR 試薬にすぐにアクセスできるように支援することを目指しています。
PCR
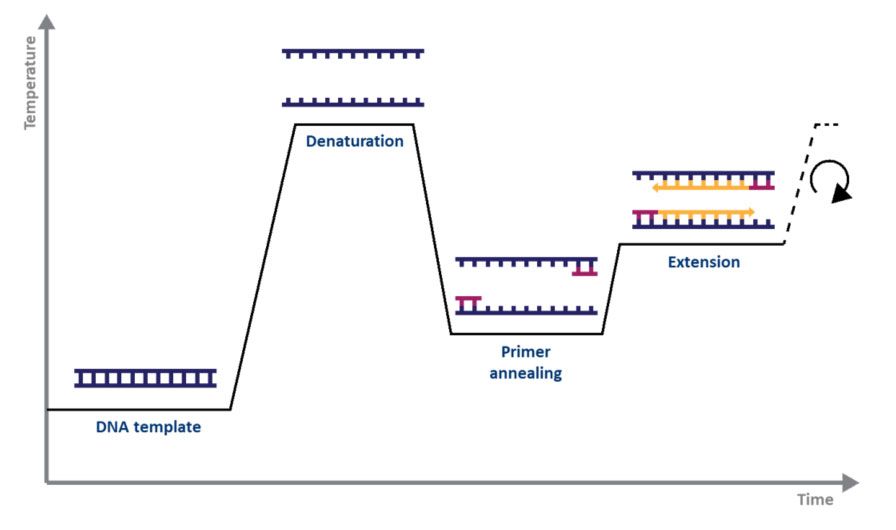
標準的な PCR の場合、必要なのは DNA ポリメラーゼ、マグネシウム、ヌクレオチド、プライマー、増幅する DNA テンプレート、およびサーモサイクラーだけです。PCR メカニズムはその目的と同じくらい単純です。1) 二本鎖 DNA (dsDNA) が熱変性され、2) プライマーが 1 本の DNA 鎖に整列し、3) プライマーが DNA ポリメラーゼによって伸長され、その結果、元の DNA 鎖。一連の温度と時間にわたる変性、アニーリング、および伸長のプロセスは、増幅の 1 サイクルとして知られています (図 1)。
| 図1。PCRによる増幅サイクルの概略図。 |
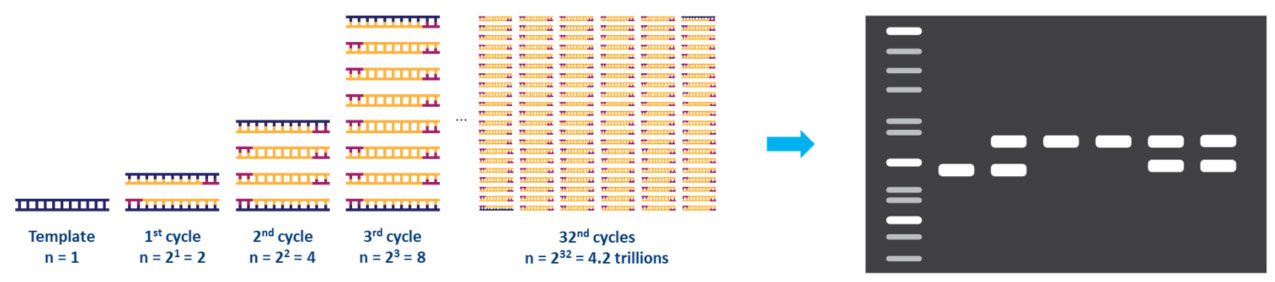
サイクルの各ステップは、使用するテンプレートとプライマーセットに合わせて最適化する必要があります。このサイクルは約 20 ~ 40 回繰り返され、増幅産物は通常はアガロースゲルによって分析できます (図 2)。
| 図2。PCRによるDNAテンプレートの増幅とアガロースゲル電気泳動による分析。 |
PCR は非常に感度の高い方法であり、1 回の反応に必要な量は非常に少ないため、複数の反応用にマスター ミックスを調製することをお勧めします。マスター ミックスはよく混合してから反応数ごとに分割し、各反応に同じ量の酵素、dNTP、およびプライマーが含まれるようにする必要があります。Enzo Life Sciences などの多くのサプライヤーは、プライマーと DNA テンプレート以外のすべてがすでに含まれている PCR ミックスも提供しています。
グアニン/シトシンリッチ (GC リッチ) 領域は、標準的な PCR 技術における課題です。GC が豊富な配列は、GC 含有量が低い配列よりも安定しています。さらに、GC に富む配列はヘアピン ループなどの二次構造を形成する傾向があります。その結果、GC リッチな二本鎖は変性段階で完全に分離することが困難になります。その結果、DNA ポリメラーゼは妨げられずに新しい鎖を合成できなくなります。変性温度を高くするとこれを改善でき、アニーリング温度を高くしてアニーリング時間を短くする方向に調整すると、GC リッチ プライマーの非特異的結合を防ぐことができます。試薬を追加すると、GC リッチ配列の増幅が強化されます。DMSO、グリセロール、ベタインは、GC 相互作用によって引き起こされる二次構造を破壊するのに役立ち、それによって二本鎖の分離を促進します。
ホットスタート PCR
非特異的増幅は、PCR 中に発生する可能性のある問題です。PCR に使用されるほとんどの DNA ポリメラーゼは、約 68°C ~ 72°C の温度で最もよく機能します。ただし、酵素は程度は低いものの、より低い温度でも活性化する可能性があります。アニーリング温度よりもはるかに低い温度では、反応を氷上で行った場合でも、プライマーが非特異的に結合し、非特異的な増幅を引き起こす可能性があります。これは、特定の温度に達したときにのみ DNA ポリメラーゼから解離するポリメラーゼ阻害剤を使用することで防止できます。そのため、ホット スタート PCR という用語が付けられています。阻害剤は、ポリメラーゼに結合し、初期変性温度 (通常 95℃) で変性する抗体であり得ます。
高忠実度ポリメラーゼ
DNA ポリメラーゼは元のテンプレート配列に合わせてかなり正確に増幅しますが、ヌクレオチドのマッチングで間違いが発生する可能性があります。クローニングなどのアプリケーションでミスマッチが発生すると、転写産物が切断され、下流にタンパク質の誤翻訳または不活性化が生じる可能性があります。これらの不一致を回避するために、「校正」活性を持つポリメラーゼが特定され、ワークフローに組み込まれています。最初の校正ポリメラーゼである Pfu は、1991 年に Pyrococcus furiosus で同定されました。この Pfu 酵素は、3' から 5' へのエキソヌクレアーゼ活性を持っています。DNA が増幅されると、エキソヌクレアーゼは鎖の 3' 末端にあるミスマッチのヌクレオチドを除去します。その後、正しいヌクレオチドが置き換えられ、DNA 合成が続行されます。間違ったヌクレオチド配列の同定は、酵素と正しいヌクレオシド三リン酸との結合親和性に基づいており、結合効率が低いと合成が遅くなり、正しい置換が可能になります。Pfu ポリメラーゼの校正活性により、Taq DNA ポリメラーゼと比較して最終配列のエラーが少なくなります。近年、他の校正酵素が特定され、DNA 増幅中のエラー率をさらに下げるために元の Pfu 酵素に修正が加えられています。
RT-PCR
逆転写 PCR (RT-PCR) では、RNA を鋳型として使用できます。追加のステップにより、RNA の検出と増幅が可能になります。RNA は、逆転写酵素を使用して相補 DNA (cDNA) に逆転写されます。RT-PCR を成功させるには、RNA テンプレートの品質と純度が不可欠です。RT-PCR の最初のステップは、DNA/RNA ハイブリッドの合成です。逆転写酵素には、ハイブリッドの RNA 部分を分解する RNase H 機能もあります。次に、一本鎖 DNA 分子は、cDNA への逆転写酵素の DNA 依存性 DNA ポリメラーゼ活性によって完成します。ファーストストランド反応の効率は増幅プロセスに影響を与える可能性があります。ここからは、標準的な PCR 手順を使用して cDNA を増幅します。RT-PCR によって RNA を cDNA に戻すことができることには多くの利点があり、主に遺伝子発現解析に使用されます。RNA は一本鎖で非常に不安定なので、扱うのが困難です。これは一般に、生体サンプル中の RNA 転写物を定量する qPCR の最初のステップとして機能します。
qPCR および RT-qPCR
定量的 PCR (qPCR) は、さまざまな用途で核酸を検出、特徴づけ、定量するために使用されます。RT-qPCR では、多くの場合、RNA 転写物は、前述のように、最初に cDNA に逆転写することによって定量され、その後、qPCR が実行されます。標準的な PCR と同様に、DNA は変性、アニーリング、伸長という 3 つのステップを繰り返すことによって増幅されます。ただし、qPCR では、蛍光標識により PCR の進行に応じてデータを収集できます。この手法には、さまざまな方法と化学反応が利用できるため、多くの利点があります。
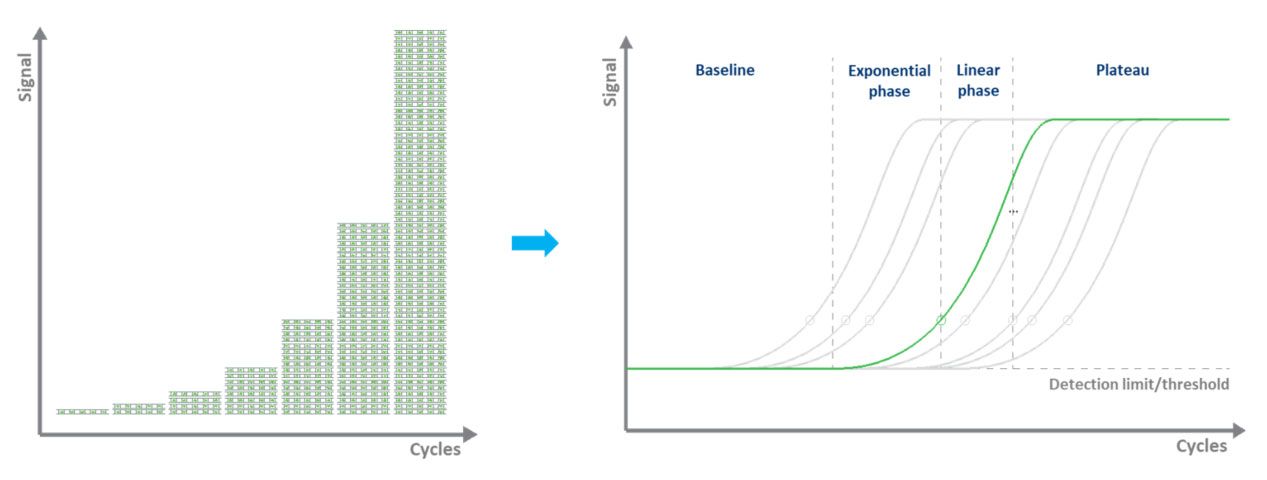
色素ベースの qPCR (通常は緑色) では、dsDNA 結合色素の使用により、蛍光標識により増幅された DNA 分子の定量が可能になります。各サイクル中に蛍光が測定されます。蛍光シグナルは複製された DNA の量に比例して増加します。したがって、DNA は「リアルタイム」で定量化されます (図 3)。色素ベースの qPCR の欠点は、一度に検査できる標的が 1 つだけであることと、色素がサンプル中に存在する任意の ds-DNA に結合することです。
| 図3.qPCR によって DNA テンプレートを増幅し、リアルタイムで蛍光シグナルを測定します。 |
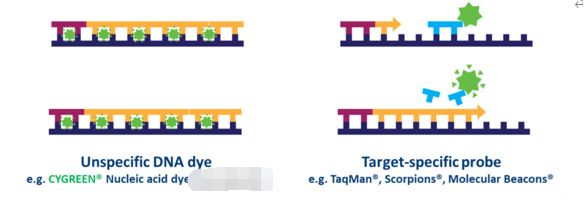
プローブベースの qPCR では、各サンプルで多くのターゲットを同時に検出できますが、これにはプライマーに加えて使用されるターゲット特異的プローブの最適化と設計が必要です。いくつかのタイプのプローブ設計が利用可能ですが、最も一般的なタイプは、フルオロフォアとクエンチャーを組み込んだ加水分解プローブです。蛍光共鳴エネルギー移動 (FRET) は、プローブが無傷である間、クエンチャーを介した蛍光色素の放出を防ぎます。ただし、PCR 反応中、プローブはプライマーの伸長と結合している特定の配列の増幅中に加水分解されます。プローブの切断により蛍光色素が消光物質から分離され、増幅に依存して蛍光が増加します (図 4)。したがって、プローブベースの qPCR 反応からの蛍光シグナルは、サンプル中に存在するプローブ標的配列の量に比例します。プローブベースの qPCR は色素ベースの qPCR よりも特異的であるため、qPCR ベースの診断アッセイでよく使用される技術です。
| 図4.色素ベースの qPCR とプローブベースの qPCR の違い。 |
等温増幅
上記の PCR 技術では、変性、アニーリング、および伸長のステップでチャンバー温度を正確に増減させるための高価なサーモサイクリング装置が必要です。このような精密な装置を必要とせず、単純なウォーターバス内で、または対象の細胞内でさえ実行できる、多くの技術が開発されています。これらの技術は総称して等温増幅と呼ばれ、指数関数的、線形、またはカスケード増幅に基づいて機能します。
等温増幅の最もよく知られたタイプは、ループ媒介等温増幅 (LAMP) です。LAMP は、65℃で指数関数的増幅を使用してテンプレート DNA または RNA を増幅します。LAMP を実行する場合、ターゲット DNA の領域に相補的な 4 ~ 6 個のプライマーを DNA ポリメラーゼとともに使用して、新しい DNA を合成します。これらのプライマーのうち 2 つは、他のプライマーの配列を認識して結合する相補的配列を持っており、新たに合成された DNA に「ループ」構造が形成されることを可能にし、その後の増幅ラウンドでのプライマーのアニーリングを助けます。LAMP は、蛍光、アガロースゲル電気泳動、比色分析などの複数の方法で視覚化できます。比色分析による生成物の有無の視覚化と検出が容易で、高価な機器が必要ないため、LAMP は臨床検査が容易に利用できない地域やサンプルの保管と輸送が困難な地域での SARS-CoV-2 検査に適した選択肢となりました。あるいは、これまで PCR サーモサイクリング装置がなかった研究室では実現できませんでした。
投稿日時: 2023 年 8 月 19 日